ゲノムワイド関連解析
ゲノム多型データは、SNPsチップや次世代シーケンサーによる変異callデータなどから得られます。このデータを利用してゲノムワイド関連解析(GWAS)を実施することができます。
Quality control解析
偽陽性を削減するためにquality control解析を行います。下記の基本解析に加えてデータごとに必要な解析を行います。
- 1.遺伝子型一致割合による血縁関係の同定
- 2.近交係数とrare allele数による適合基準の確認
- 3.性別の同定
- 4.Minor allele frequency (MAF)によるSNPsの足切り
- 5.Hardy-Weinberg平衡検定によるSNPs graphの確認と不良call SNPsの同定
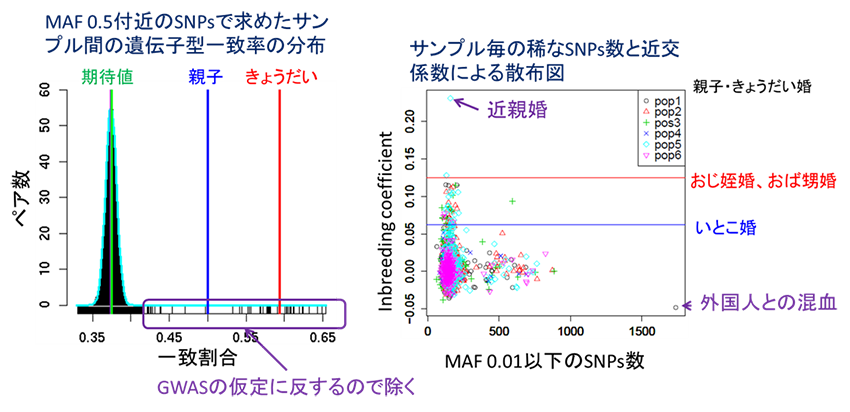
遺伝子型一致割合による血縁関係の同定(左) 近交係数とrare allele数による適合基準の確認(右)
性別の同定
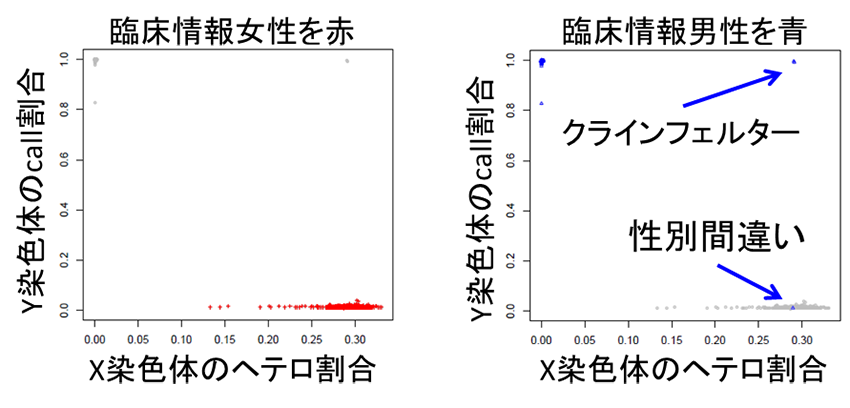
- *弊社の解析経験では、200人に一人くらいの割合で実際の性別と解析結果の性別が異なることがあります。
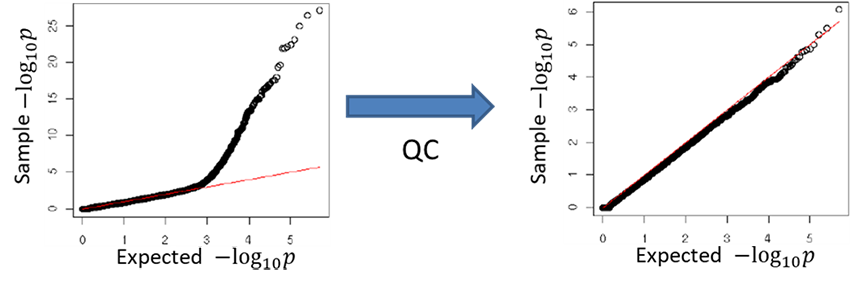
Hardy-Weinberg平衡検定のQ-Q plot

集団の構造化解析
関連を解析する集団間に分集団の不均衡が存在すると、分集団に特徴的なSNPsを多数検出してしまいます。そのため分集団の存在をチェックし、帰無仮説がどの程度の精度で成立しているのかを予め明らかにしておく必要があります。
EIGENSTRAT (Price et al. 2006)
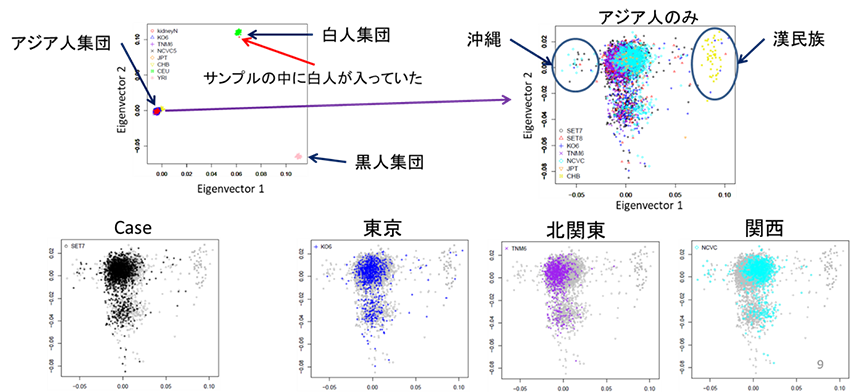
遺伝子型のCochran-Armitage trend testを行い、control集団別に偽陽性の量を推定
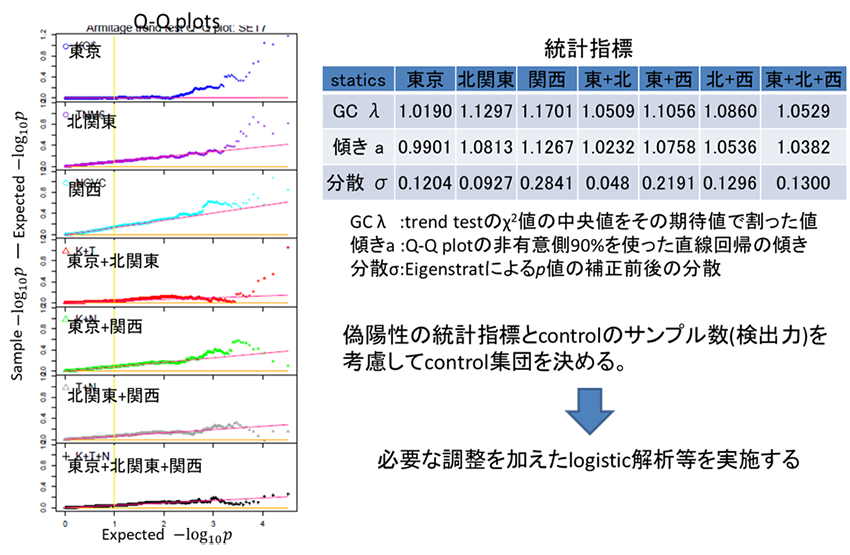
関連解析
- 遺伝子型trend test (exact)
- Logistic回帰(バイアス調整)
- Matched case-control study
- Haplotype推定を用いた関連解析
コピー数推定
IlluminaのSNPsチップをaCGHデータとしてcopy number variation (CNV)を探索することができます。
Quality control解析
プローブの選択
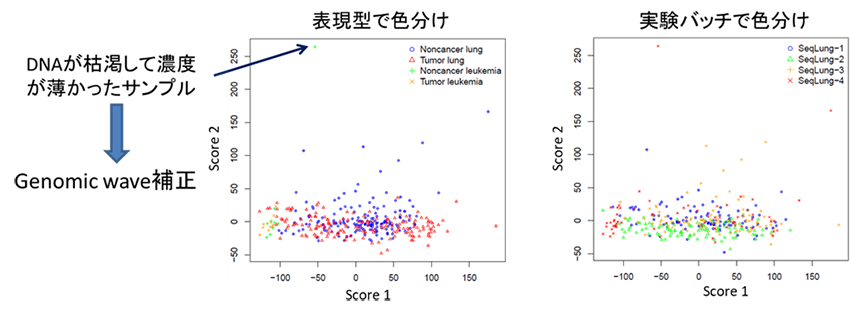
logR Ratio値の分散共分散行列を用いた主成分分析
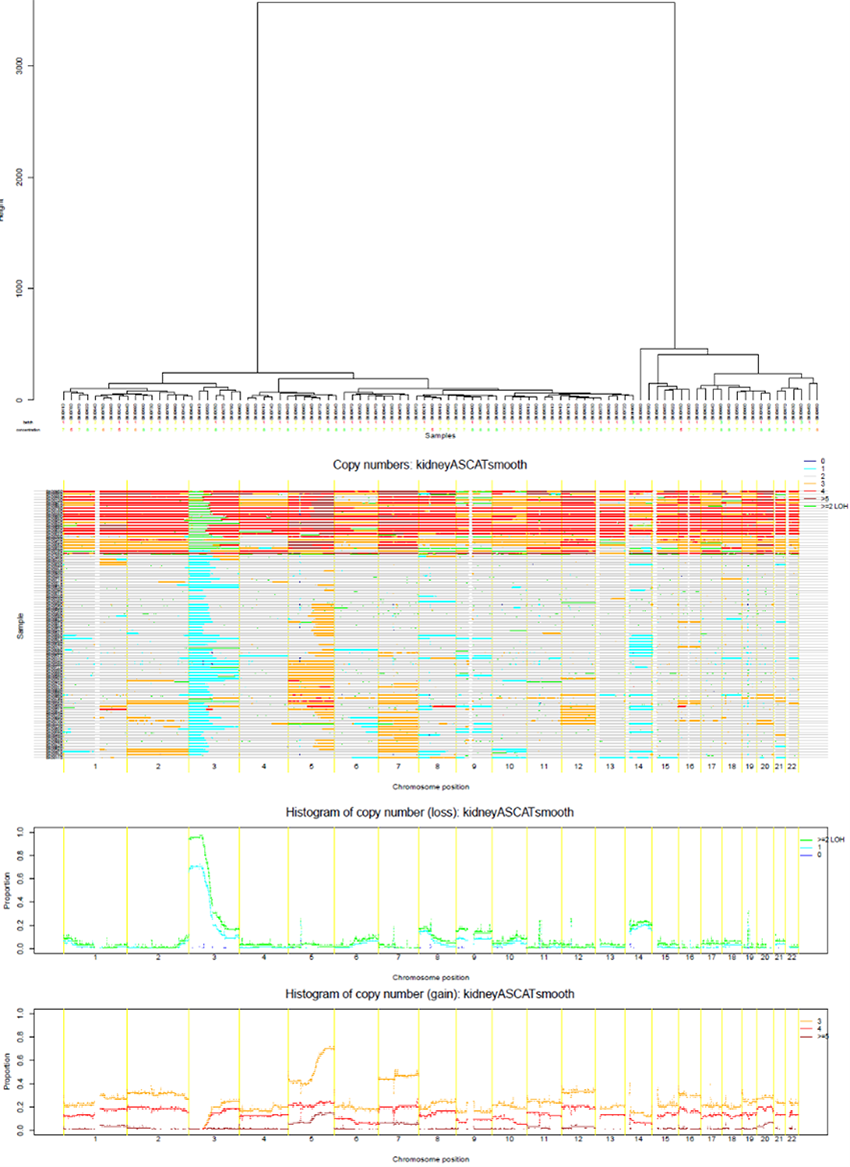
体細胞コピー数変異推定
多くのがん細胞ではある程度大域的な染色体コピー数異常が生じており、がん種毎に特徴的な増幅や欠失が見られることがあります。
体細胞コピー数変異推定結果をクラスタリングして増幅、欠失のヒストグラムと共に表示
参考書籍: がんゲノムデータ解析 第4章がんのCNV検出
https://www.medsi.co.jp/products/detail/3866
技術調査
弊社が作成した技術調査報告書例を下記に載せました。
- cDNAリファレンスデータの統合(PDF/101KB)
- 全エクソンシーケンシングデータからの生殖細胞系ゲノムコピー数予測ソフトウエアの調査(PDF/3,500KB)
- 核酸omicsデータの品質管理解析(PDF/2,713KB)
- Illumina Omni チップデータからの体細胞ゲノムコピー数異常予測ソフトウエアの調査(PDF/692KB)