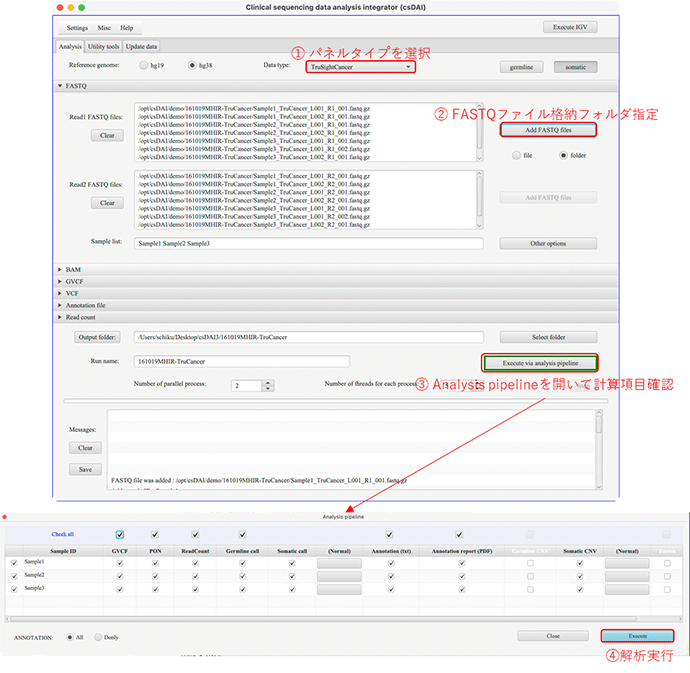
通常NGSから出力されるreadデータ(FASTQファイル)からアノテーション情報の付与までを行うにはバイオインフォマティクスやLinuxの知識が必要となります。csDAI®ではGUIからFASTQファイルを指定して解析を実行するだけでエキスパートパネルに提供するアノテーションレポートを作成することができます。
解析の実行
- ①パネルタイプを選択
- ②FASTQファイルが格納されているフォルダーを指定(NGSランのフォルダーを選択)
- ③Analysis pipelineで計算項目を確認
- ④実行(Executeボタン)
Analysis pipelineの選択項目。G:germline、S:somatic、R:RNA-seq。〇は選択可能、△は非推奨、×は選択不可。
左右スクロールで表全体を閲覧できます
| 項目 | G | S | R | 計算内容 |
|---|---|---|---|---|
|
GVCF |
〇 |
〇 |
〇 |
Germline callを行うためのGVCF計算。 |
|
PON |
〇 |
△ |
× |
Mutect2計算で指定するpanel of normal VCFファイルを出力。がんサンプルでも計算できるがPONの統合には含めてはいけない。 |
|
ReadCount |
〇 |
〇 |
× |
コピー数解析のためのread countファイルを出力。 |
|
Germline call |
〇 |
〇 |
〇 |
Germline callを行い、VCFファイルを出力。 |
|
Somatic call |
× |
〇 |
△ |
Mutect2によるsomatic callを実行。Normalを指定することを推奨。 |
|
Annotation(txt) |
〇 |
〇 |
〇 |
Germline call後にアノテーションファイルを出力。 |
|
Annotation report |
〇 |
〇 |
〇 |
アノテーションファイルからサンプル毎のアノテーションレポートファイルを出力。 |
|
Germline CNV |
〇 |
× |
× |
Germline CNV解析を実行。 |
|
Somatic CNV |
× |
〇 |
× |
Somatic CNV解析を実行。Normalの指定が必要。 |
|
Fusion |
× |
× |
〇 |
融合遺伝子解析を実行。 |
|
Expression |
× |
× |
〇 |
RNA発現量解析を実行。 |
出力
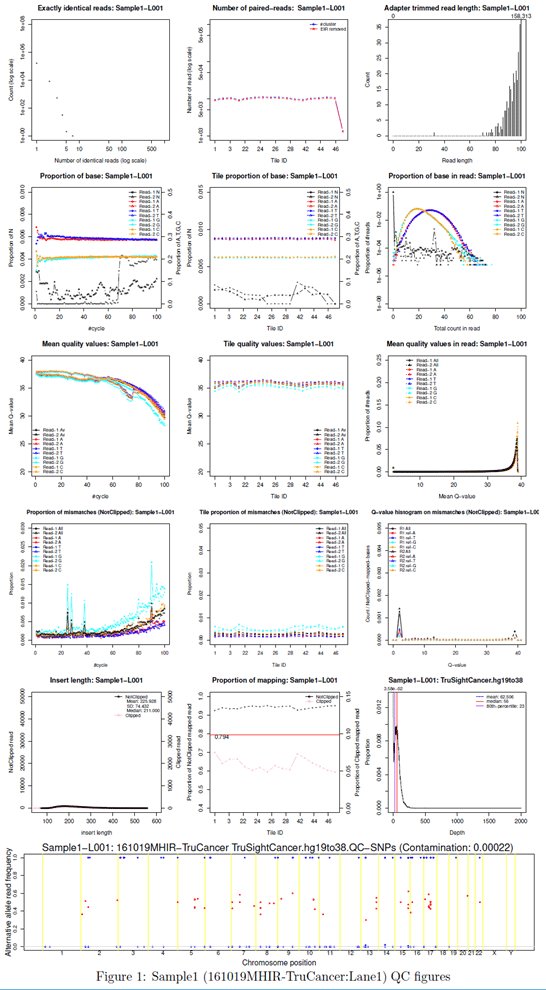
- QCレポート(text, PDF)
- アノテーションレポート(text, PDF)
- CNVアノテーション(text) (DNA-seqのみ)
- Fusion/Expression(text, PDF)(RNA-seqのみ)
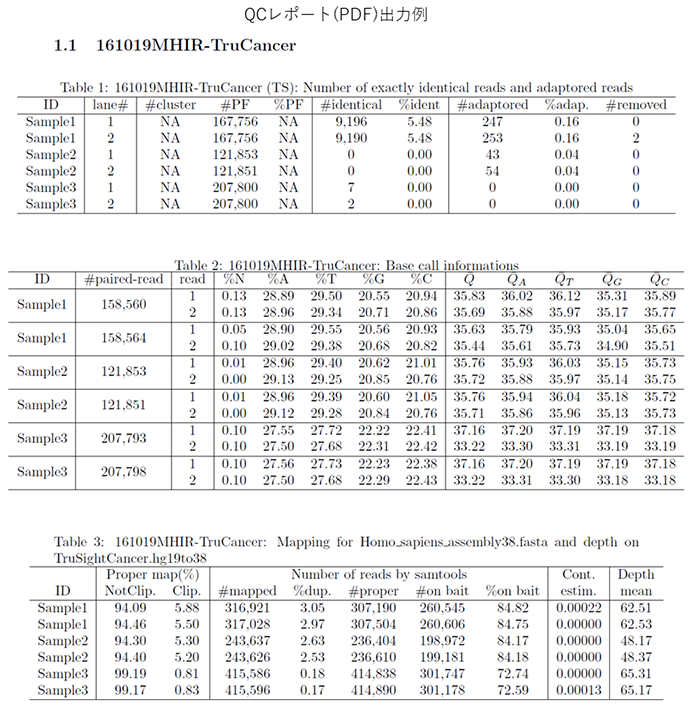
QCレポート出力例
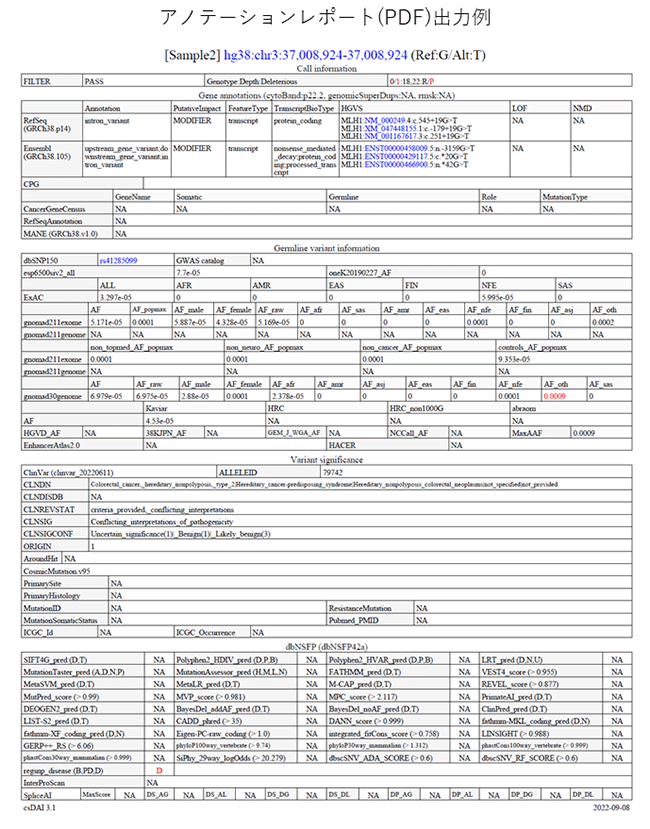
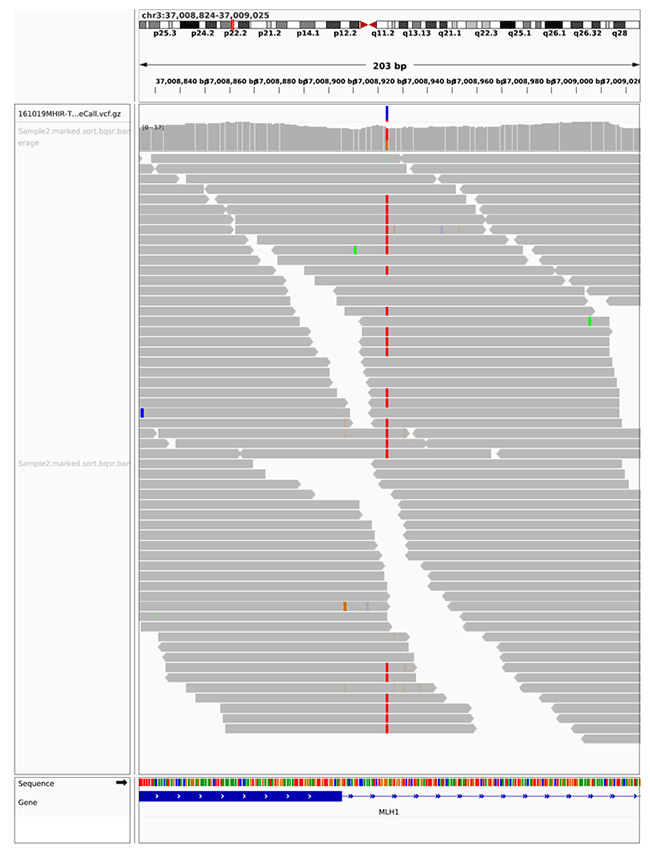
アノテーションレポート出力例