オミックスデータ解析 トランスクリプトームデータ
- サービスの概要
- ゲノム多型データ
- メチレーションデータ
- トランスクリプトームデータ
- メタボロームデータ
- 多層オミックスデータ
トランスクリプトームデータにはDNAマイクロアレイによるmRNAの発現量データに加え、miRNA用チップデータ、次世代シーケンサーによるRNA-seqデータからの推定量などがあります。
Quality control解析
実験データの性質を把握するためにデータの可視化を実施します。実験データには、測定プローブのロットによる差、測定機器の個体差や実験実施者の違い、実験日(例えば気温や湿度)の差等の無数の原因からバイアスが発生する可能性があります。またヒューマンエラー(ラベルのつけ間違い等)も起こるため、これらのチェックを実施することは、解析を実施する前の重要な作業になります。下記のような手法を用いてデータの品質を確認します。
手法例:
- 発現量のヒストグラム
- ボックスプロット
- 主成分分析
例:mRNAの発現量データの主成分分析
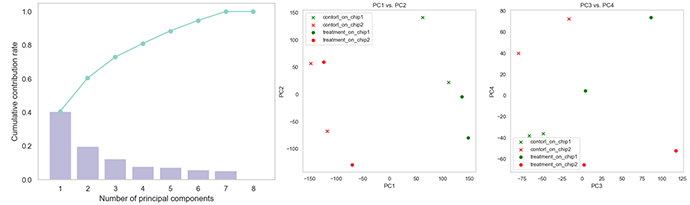
処置群とコントロール群の差よりもチップバイアスが大きかった例。
統計解析を行う場合にはチップバイアスを考慮する必要が生じる。
遺伝子(プローブ)毎の統計解析・多変量解析
2群以上のデータに対して各プローブ・遺伝子の有意差を評価します。検体数やデータの分散から適切な手法を選定して実施します。
左右スクロールで表全体を閲覧できます
| 解析手法 | 詳細手法 |
|---|---|
| ノンパラメトリック検定 | Kruskal-Wallis検定、Wilcoxon検定 |
| パラメトリック検定 | 分散分析、Studentのt検定、 Welchのt検定 |
| 傾向性検定 | Jonckheere-Terpstra検定、線形回帰モデルを用いた検定 |
| モデルを用いた解析 | 尤度比検定、ロジステック回帰、一般化線形モデル |
| 多変量解析 | 主成分分析、クラスター分析、因子分析、判別分析 |
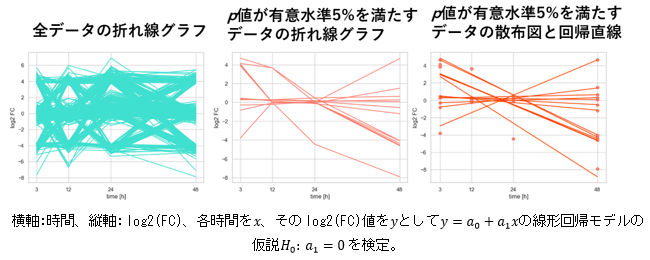
例: miRNAの発現量データの傾向性検定
パスウェイ変動解析
2群間のデータに対して遺伝子セット単位で有意差を評価します。データの確認や考察のために解析結果には遺伝子セットの説明やリンク等もまとめてスプレッドシート形式で整理します。
例:Gene Set Enrichment Analysis(GSEA)*1を用いた解析
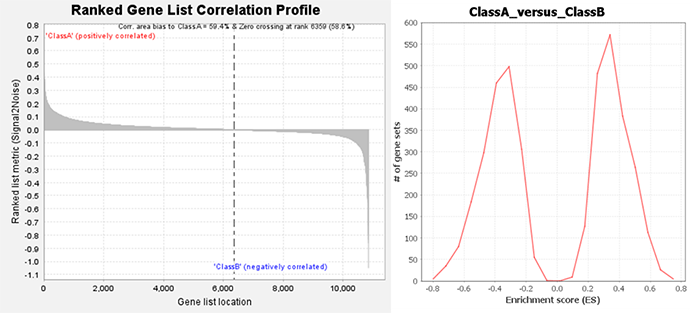
- *1Subramanian, Aravind, et al. Proceedings of the National Academy of Sciences 102.43 (2005): 15545-15550.
時系列データ解析
遺伝子発現の時系列データから遺伝子発現量の変化を解析することにより、様々な相関やクラスタリング等の解析を行います。転写因子やリン酸化酵素などの機能情報を考量した解析を行うこともできます。
例:時間帯毎の毒性と遺伝子発現量の相関ランキング*2
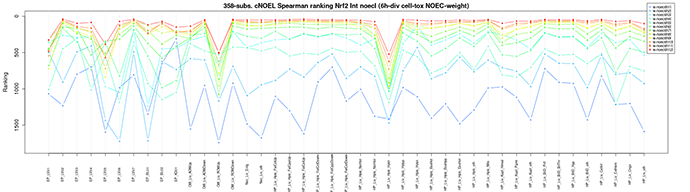
横軸:各種有害性項目、縦軸:in silico記述子中における遺伝子発現量の相関ランキング
- *2日本動物実験代替法学会第34回大会(2021年)発表資料より抜粋
